研究室の紹介Laboratory
糖鎖立体構造
糖鎖は単糖がグリコシド結合により結合しているが、その自由度の高さから多様なコンフォメーションが可能なため、立体構造を決定することは困難です。しかし、Protein Data Bank (PDB)に登録されているエントリーには糖鎖を含むことがあり、糖鎖の立体構造を理解するための情報として有用と考えられます。そして、糖タンパク質においては糖鎖が結合しているアミノ酸残基の情報や、糖鎖を認識するタンパク質であるレクチンと糖鎖の立体的な位置関係の情報などを得ることができます。我々はこれらの情報を解析するためのツールとして、PDB2Glycanを開発しました。このツールは糖鎖を含むPDBのエントリーを解析し、糖鎖構造を抽出します。その糖鎖データをもとに上述の情報や糖鎖近傍のアミノ酸残基情報などを解析します。また、糖鎖構造は我々の開発した線形文字列表記法であるWURCSに変換するとともに、国際糖鎖構造リポジトリGlyTouCanのアクセッション番号を付与します。そして、これらの情報をリンクトオープンデータ(LOD)として出力します。PDBのデータ更新は毎週実施されるため、このツールによる解析も毎週実施しています。そして、この生成したLODを利用してGlycoNAVIのデータベースとしてTCarp[1]として公開しています。 一方、我々はPDBに登録されている全エントリーに対して糖鎖分子の表現の改善(Carbohydrate Remediation)[2]を、PDBグループと協働し実施しました[3]。これにより、PDBの登録システムであるOneDepに我々のツールを組み込み、PDBにより糖鎖とアノテーションされた分子にWURCSを付与したデータがPDBの公式データとして公開されるようになりました[4]。WURCSが組み込まれたことにより、PDBのデータとGlyTouCanのアクセッション番号を関連付けつことが容易になりました。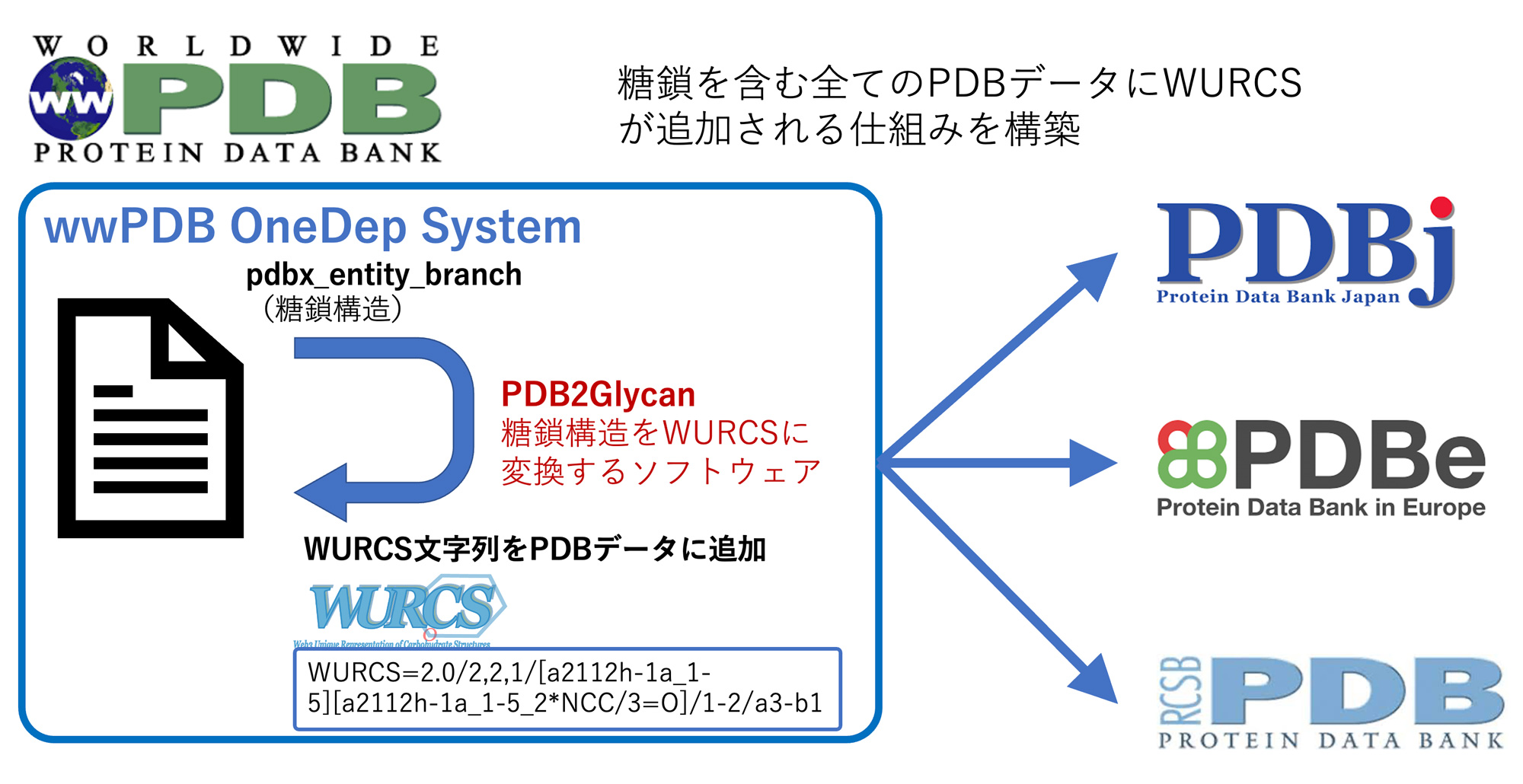
参考文献
- https://glyconavi.org/TCarp/
- https://www.wwpdb.org/documentation/carbohydrate-remediation
- https://pdbj.org/news/20200729?lang=ja
- Enhanced validation of small-molecule ligands and carbohydrates in the Protein Data Bank. Feng Z, Westbrook JD, Sala R, Smart OS, Bricogne G, Matsubara M, Yamada I, Tsuchiya S, Aoki-Kinoshita KF, Hoch JC, Kurisu G, Velankar S, Burley SK, Young JY., Structure. 2021 Apr 1;29(4):393-400.e1. doi: 10.1016/j.str.2021.02.004. Epub 2021 Mar 2. PMID:33657417
